微生物所研究团队系统挖掘并建立模式甲烷古菌遗传元件库

近日,中国科学院微生物研究所东秀珠研究团队在模式甲烷古菌的遗传元件库挖掘方面取得新进展,研究成果发表于Metabolic Engineering期刊。
模式古菌物种——海沼甲烷球菌,因其可以利用CO2/H2快速生长及高效产甲烷和萜类化合物前体等,近年在开发利用方面引起关注。但由于标准化遗传元件的匮乏,极大限制了其作为代表物种进行古菌基本生物学及生物技术开发的研究。
东秀珠团队在前期开发该模式古菌高效基因编辑工具 (Microbiol Spectr, 2021)及建立组学水平元件挖掘技术dRNA-seq及Term-seq (PLoS Genet, 2019; Nucleic Acids Res, 2020)的基础上,系统挖掘了海沼甲烷球菌的遗传元件;对转录元件(启动子)和翻译元件(RBS)进行了标准化分析,且通过扫描突变等,揭示了古菌强启动子和强RBS元件的关键碱基组成规律;成功改造了弱启动子和弱RBS,使其转录和翻译活性分别提升了120倍和3倍。由此,建立了海沼甲烷球菌的标准化遗传元件库,可实现在转录和翻译水平的动态调控范围分别可达104倍和100倍。利用这些标准化遗传元件,大幅提升了海沼甲烷球菌的蛋白表达能力(41倍);且实现了对必需基因的成功敲低。同时,筛选得到了8个染色体中性整合位点,开发了CRISPR-Cas9敲入的一步染色体markerless整合策略,保证了外源基因及代谢途径的稳定表达。因此,该研究扩充了海沼甲烷球菌的标准化遗传元件库及遗传工具,推动了其做为古菌底盘进行古菌生物学研究及CO2固定的合成生物学研究;揭示了古菌遗传元件组成规律并实现成功改造,提出了高效蛋白表达系统及代谢途径/基因调控的新思路。
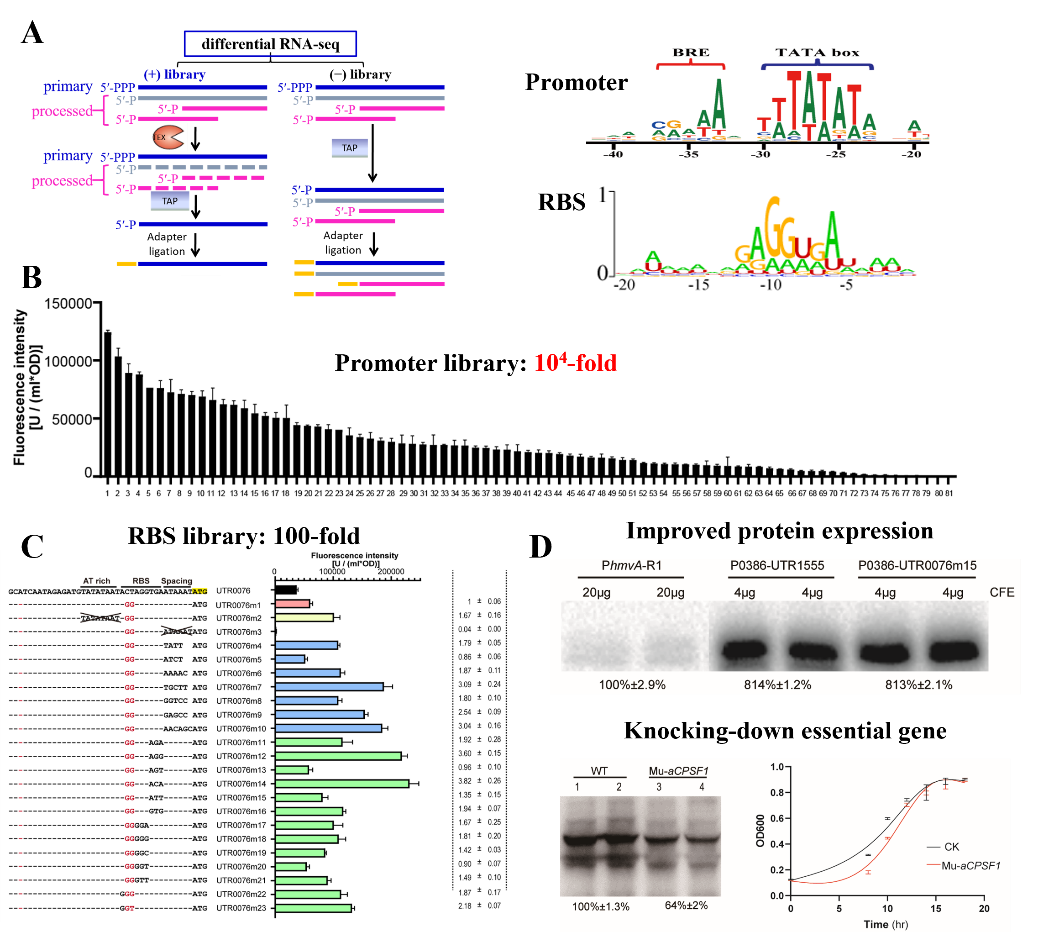
模式甲烷古菌遗传元件库的挖掘及应用
中国科学院微生物所与天津大学联合培养硕士生徐晴为文章第一作者,李洁副研究员为本文通讯作者。该研究得到国家自然科学基金项目及国家重点研发计划项目支持。
论文链接:https://www.sciencedirect.com/science/article/pii/S1096717623001143





